Chi Cinamomum, với khoảng 250 loài đã được ghi nhận trên thế giới, là một nhóm thực vật có tính ứng dụng cao trong y học. Tại Việt Nam, mặc dù các nghiên cứu trước đây đã xác định được 3 loài quế chính (Cinnamomum cassia, Cinnamomum loureirii, Cinnamomum zeylanicum), tuy nhiên sự phân loại này vẫn còn nhiều tranh cãi do chủ yếu dựa trên các đặc điểm hình thái và hóa học. Để làm rõ hơn về đa dạng di truyền của loài quế tại Việt Nam, chúng tôi đã tiến hành nghiên cứu trình tự đoạn xen kẽ trnL/trnF để đánh giá trên 18 mẫu quế thu thập từ bốn vùng sinh thái khác nhau trên khắp Việt Nam (Đông Bắc, Tây Bắc, Bắc Trung Bộ và Duyên hải miền Trung). Các mẫu được chọn là những cây quế trưởng thành (10-15 tuổi), đại diện cho sự đa dạng địa lý của loài quế tại Việt Nam. Kết quả phân tích trình tự nucleotide vùng TrnL/TrnF đã cung cấp một cái nhìn sâu hơn về mối quan hệ di truyền của các mẫu. Mức độ tương đồng di truyền cao giữa các mẫu, với tỷ lệ A+T trung bình là 63,2% và G+C là 36,8%. Hệ số tương đồng trình tự giữa các mẫu dao động từ 98,91% đến 100%, cùng với kết quả so sánh với các dữ liệu trong ngân hàng gen, đã xác nhận tất cả các mẫu đều thuộc loài Cinnamomum cassia.
1. Đặt vấn đề
Quế là một loại dược liệu quý được sử dụng từ hàng nghìn năm về trước trong các nền Y học cổ truyền ở Việt Nam, Trung Quốc…, để trị phong thấp, bong gân, viêm phế quản, ăn không tiêu, đau cơ, tiêu chảy, rối loạn kinh nguyệt, cảm lạnh,… [1], [2]. Nó cũng là một loại gia vị truyền thống, được sử dụng rộng rãi trên khắp thế giới; phụ gia cho các ngành công nghiệp, nguyên liệu sản xuất mỹ phẩm, dược phẩm, lấy gỗ, chất bảo quản thực phẩm, thuốc trừ sâu, trừ nấm có nguồn gốc thực vật [3], [4]. Năm 2021, Việt Nam là quốc gia dẫn đầu trên thế giới về diện tích trồng Quế và xuất khẩu với giá trị trên 200 triệu USD [5]. Việc phát triển mạnh trồng Quế trong khoảng 20 năm gần đây do việc thay đổi mô hình trồng, từ trồng thưa sang trồng mật độ cao và cắt tỉa để chưng cất tinh dầu cành con và lá, từ đó nâng cao thu nhập từ trồng quế cũng như có thu nhập ngay từ năm thứ tư sau khi trồng. Việc phát triển mạnh mẽ này đặt ra nhiều thách thức cho sự phát triển bền vững và chất lượng các sản phẩm từ cây quế ở Việt Nam [6]. Sự phát triển ồ ạt trong giai đoạn gần đây đã khiến cho các vùng trồng quế có sự xáo trộn về thành phần loài, gây giảm chất lượng đầu ra của một số vùng trồng quế bản địa như Thanh Hóa, Trà My.
Nghiên cứu đánh giá mối quan hệ di truyền giữa 8 loài thuộc chi Cinnamomum ở Nam Ấn Độ dựa trên chỉ thị RAPD, tác giả đã thành công trong việc phân loại và nghiên cứu dựa trên cây phát sinh loài ở các loài thuộc chi Cinnamomum. Kết quả đã thu được 111 băng ADN trong đó có 98% băng đa hình. Kích thước băng nhân lên được cao nhất là 4200 bp và thấp nhất là 100bp, hệ số tương đồng di truyền dao động từ 0,17 (giữa C.zeylanicum và C.travancoricum) đến 0,81 (giữa C.macrocarpum và C.travancoricum). Dựa vào sơ đồ hình cây chia thành 2 nhóm. Nhóm A gồm C. malabatrum, C. sulphuratum, C. macrocarpum, C. travancoricum, C. wightii, C. nicolsonianum và C. walaiwarense. Nhóm B chỉ có loài C.zeylanicum [7]. Năm 2009, Pushpa và cộng sự, sử dụng phương pháp phân tích trình tự DNA để nghiên cứu sự đa dạng di truyền của các loài quế (Cinnamomum) ở Sri Lanka nhận thấy vùng ITS đa dạng nhất với với 7 vị trí đa hình được tìm thấy trong số 9 loài Quế. Băng phân tích trình tự nucleotide của các vùng cpDNA khác nhau và một vùng ITS của rDNA. Các vùng cpDNA được nghiên cứu là các đoạn mã gen giữa trnL-trnF, trnT-trnL, trnH-psbA và intron trnL. Trong số sử dụng 14 mồi; vùng trnL lục lạp 6 mồi 3 cặp cho trnL intron và trnL-trnF IGS liền kề (c/d, e/f và a/b), trnH-psbA spacer 2 mồi (trnH và psbA), vùng ITS có 2 mồi ITS4 và ITS5A [8]. Năm 2011, nhóm nghiên cứu Kuen – Yin Ho và cộng sự đã điều tra mối quan hệ tiến hóa giữa các loài Cinnamomum. Họ đã sử dụng kết hợp ba cách tiếp cận khác nhau; Phân tích hình thái lá, phân tích ISSR, phân tích ITS, để phân tích sự đa dạng di truyền và mối quan hệ họ hàng giữa 12 loài Cinnamomum ở Đài Loan, 7 đoạn mồi được lựa chọn ISSR đã được sử dụng để phân tích sự đa dạng di truyền trong các quần thể Cinnamomum camphora, ITS đã được sử dụng để xây dựng cây phát sinh loài của các loài Cinnamomum ở Đài Loan. Kết quả phương pháp ISSR là một phương pháp hiệu quả để phân tích sự đa dạng di truyền trong các quần thể. ITS là một phương pháp hiệu quả để xây dựng cây phát sinh loài xác xác định các loài lai [9].
Ở Việt Nam Năm 2015, Khuất Hữu Trung và cộng sự đánh giá đa dạng di truyền của quế Thanh Hóa bằng chỉ thị RAPD – PCR gồm 22 mồi ngẫu nhiên đã được sử dụng để đánh giá đa dạng di truyền 32 mẫu giống quế Thanh Hóa. Kết quả phân tích PCR nhân lên được 3,137 băng bao gồm 126 loại băng với kích cỡ khác nhau. Trong đó 61 băng đa hình (chiếm 48,41%) và 65 băng đơn hình (chiếm 51,59%). Số băng nhân lên dao động từ 3 đến 11 loại băng, trung bình 5,8 băng/mồi. Hệ số tương đồng của 32 mẫu dao động 0,67 đến 0,97. Dựa vào hệ số tương đồng chia 32 mẫu giống nghiên cứu thành 6 nhóm lớn. Kết quả nghiên cứu cũng xác định được 2 băng cá biệt ở 2 mồi có thể làm marker nhận dạng chính xác 2 nguồn gen khác nhau. Mồi OPN20 và UBC 728 có thể phân biệt mẫu giống tương ứng là TT02 vã XC24 [10]. Hiện nay, các nghiên cứu trên đối tượng cây Quế ở Việt Nam chủ yếu tập trung vào đặc điểm hình thái và phân tích đa dạng di truyền mới được thực hiện theo từng địa phương. Bởi vậy việc sử dụng kết hợp các mồi ở vùng DNA lục lạp để phân tích định danh loài [11]. Được thực hiện trên cả nước giúp đánh giá lại tình hình phân bố thực tế các loài Quế, cung cấp dữ liệu cho chọn giống, xây dựng vùng trồng, sản xuất các sản phẩm chất lượng, truy xuất được nguồn gốc rõ ràng, từ đó góp phần đưa Quế trở thành dược liệu thế mạnh và đa giá trị của Việt Nam.
Xuất phát từ thực tế nêu trên, mục tiêu nghiên cứu của chúng tôi là “Đánh giá đa dạng di truyền của một số mẫu Quế trồng ở Việt Nam bằng chỉ thị TrnL/TrnF. Sử dụng 18 mẫu lá quế được thu thập từ các tỉnh Đông Bắc, Tây Bắc, Bắc Trung Bộ và Duyên hải miền Trung. Để xác định Việt Nam có 3 loài quế như trong các sách đã ghi nhận, cung cấp dữ liệu cho chọn giống, xây dựng vùng trồng, sản xuất các sản phẩm chất lượng, truy xuất được nguồn gốc rõ ràng, từ đó góp phần đưa Quế trở thành dược liệu thế mạnh và đa giá trị của Việt Nam.
2. Vật liệu và phương pháp nghiên cứu
2.1. Vật liệu nghiên cứu
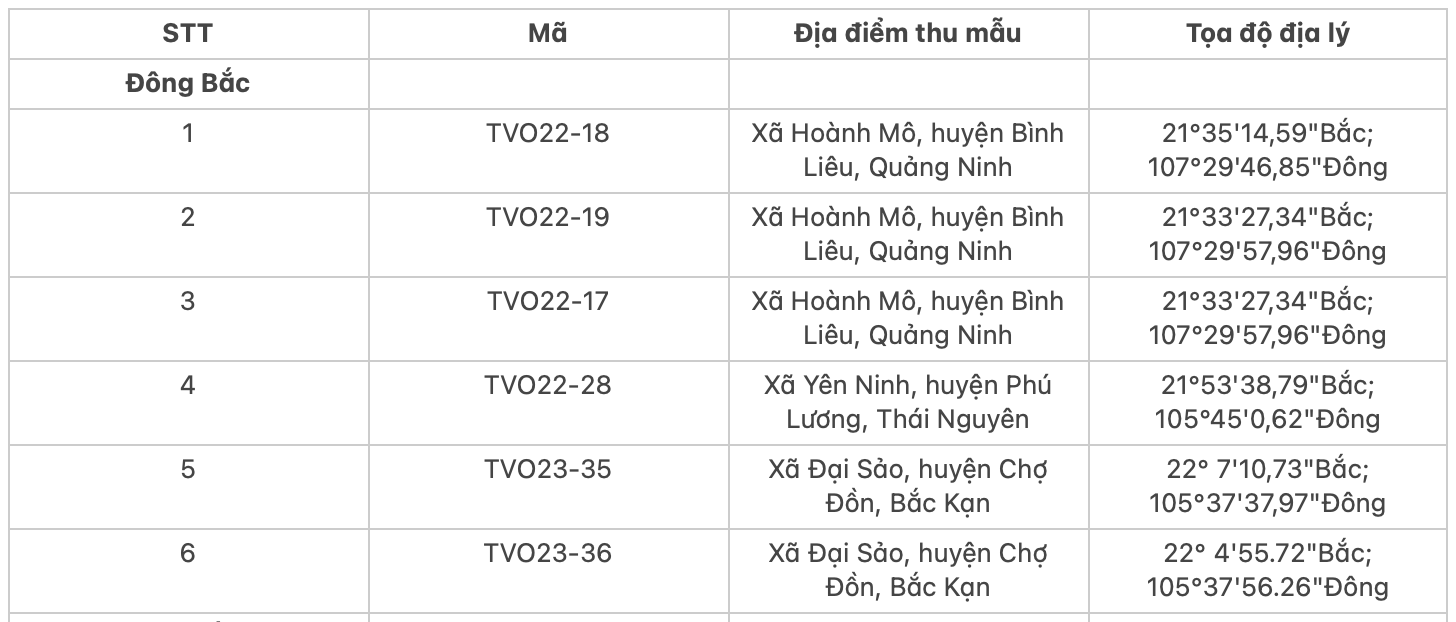
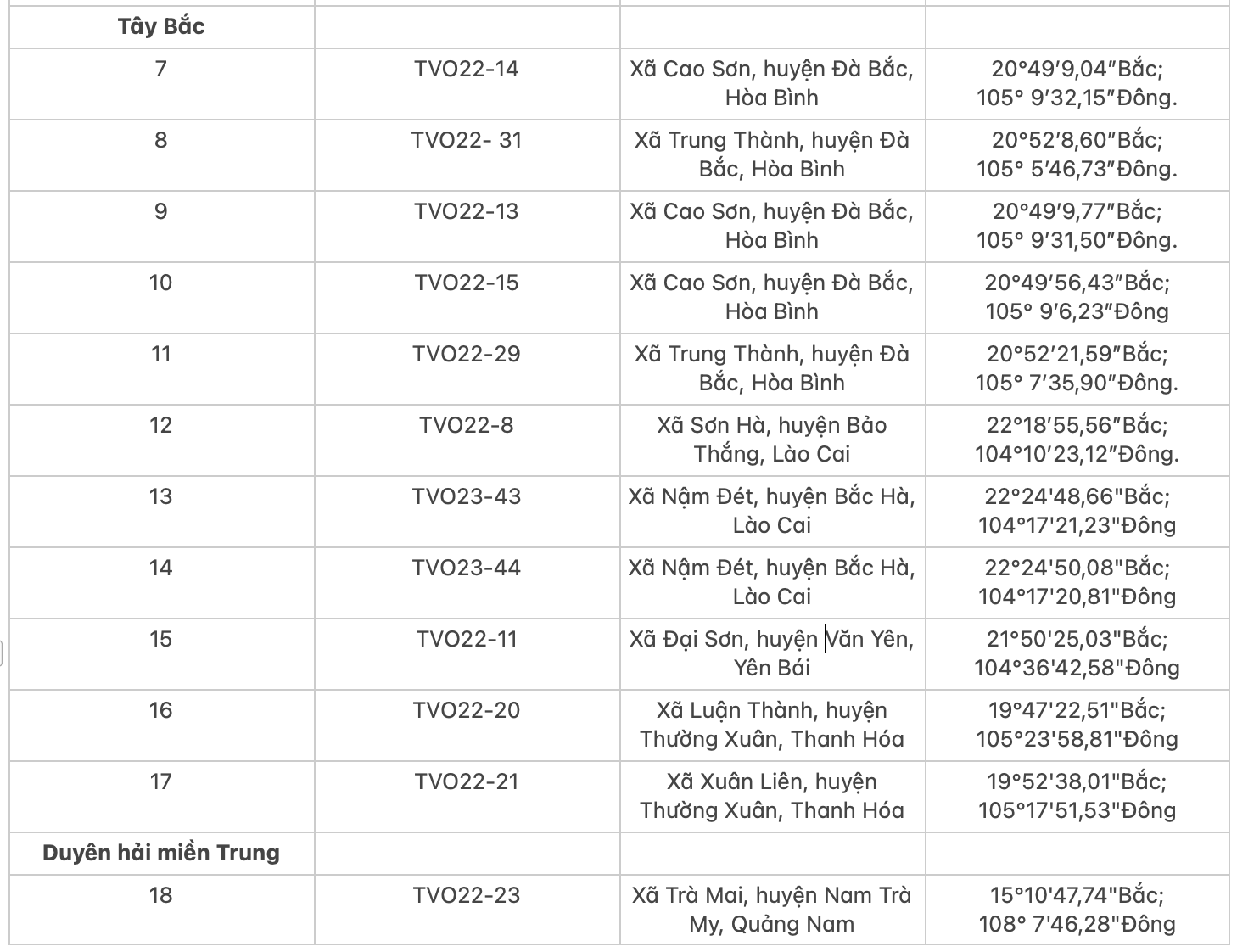
Vật liệu nghiên cứu gồm 18 mẫu lá quế bánh tẻ của các cây từ 10-15 tuổi được thu thập tại các vùng trồng trong cả nước thuộc 4 vùng sinh thái Đông Bắc (Quảng Ninh, Bắc Kạn, Thái Nguyên); Tây Bắc (Yên Bái, Lào Cai, Hòa Bình); Bắc Trung Bộ (Thanh Hóa); Duyên hải miền Trung (Quảng Nam). Các cây thu mẫu lá được ghi lại tọa độ địa lý để truy xuất khi cần thiết. Thông tin các mẫu đã thu thập được trình bày ở Bảng 1 (Bảng 1).
Bảng 1: Danh mục các mẫu quế nghiên cứu
2.2. Phương pháp nghiên cứu
2.2.1. Kết quả tách chiết ADN tổng số
Các mẫu Quế được thập được sẽ được tách ADN tổng số bằng phương pháp phương pháp tách CTAB có cải tiến của của Asif and Cannon (2005) để tiến hành tách chiết ADN từ 18 mẫu nghiên cứu [12]. Sau đó, ADN tổng số sẽ được kiểm tra độ tinh sạch bằng diện di trên gel agarose 1%. Mẫu ADN đạt yêu cầu sẽ được bảo quản trong điều kiện lạnh sâu (-20oC) cho các nghiên cứu về sau.
2.2.2. Khuếch đại vùng ITS bằng kỹ thuật PCR
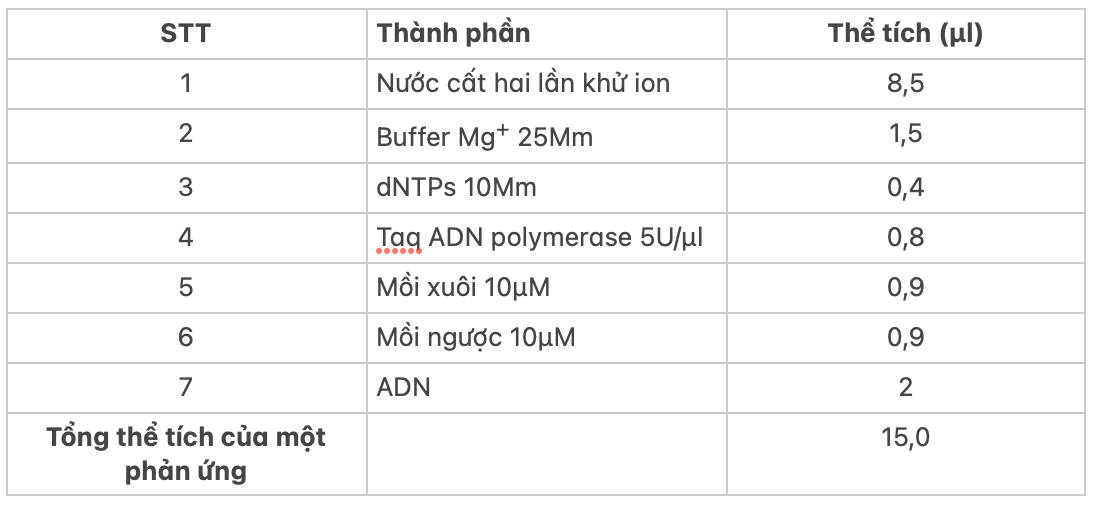
- Khuếch đại vùng TrnL/TrnF với Taq DNA pol 2x- preMix: 0.1U/mL Taq DNA Polymerase, 0.4 mM dATP, 0.4 mM dGTP, 0.4 mM dCTP, 0.4 mM dTTP, 4mM MgSO4, 20mM KCl, 16 mM (NH4)2SO4, 20 mM Tris-HCl (pH8) và cặp mồi TrnL ( 5- GGTTCAAGTCCCTCTTATCCCATCCC- 3) và TrnF (ATTTGAACTGGTGACACGAG). Thành phần phản ứng và chương trình chạy PCR được thể hiện ở bảng 2, 3 :
Bảng 2: Thành phần một phản ứng PCR
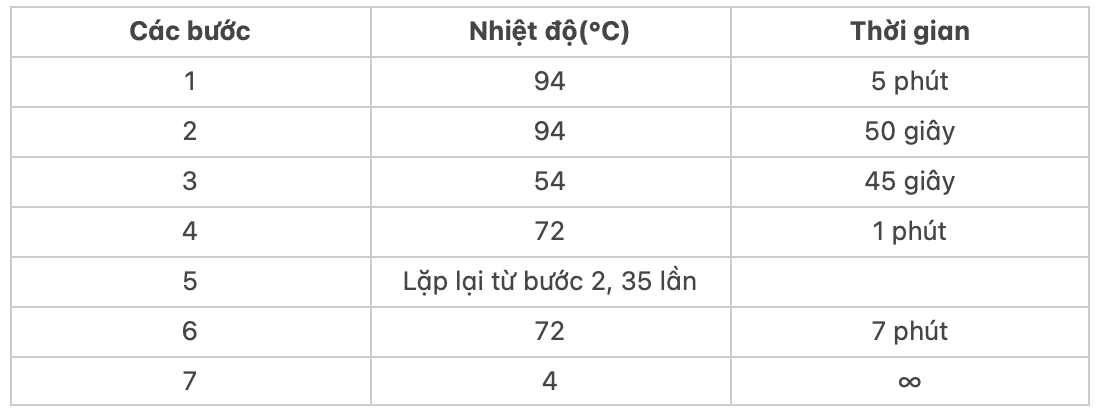
Chương trình chạy PCR: Phản ứng PCR được tiến hành trong ống eppendorf 0,2 ml và thực hiện trên máy Mastercycler epgradient S theo chu trình sau (Bảng 3).
Bảng 3: Chương trình chạy PCR
* Sản phẩm PCR được điện di cùng với thang chuẩn 100 bp (DNA ladder) trên gel agarose 1 % ở hiệu điện thế 120 V, cường độ dòng điện 65 mA trong 30 phút. Kết quả sẽ hiển thị khi xem dưới tia UV có bước sóng 365 nm. Bằng cách này, ta có thể kiểm tra sự có mặt của các băng DNA và chất lượng sản phẩm của từng mẫu.
* Sản phẩm PCR sau khi được tinh sạch, được giải trình tự tại công ty TNHH DNA Sequecing – Việt Nam. Kết quả giải trình tự được được so sánh với các trình tự tương đồng trên NCBI.
3. Kết quả và Thảo luận
3.1. Kết quả khuếch đại vùng trnL-trnF bằng PCR
Kết quả điện di sản phẩm PCR và thang chuẩn DNA trên gel agarose 1% cho thấy đoạn gene trnL-trnF đều được khuếch đại ở các mẫu ở vị trí xấp xỉ 400bp, các băng ADN thu được sáng và rõ thể hiện ở Hình 1. Kết quả này một lần nữa khẳng định chất lượng và kích thước đủ lớn cũng như không lẫn các băng phụ gây ảnh hưởng đến giải trình tự, điều đó cho thấy sản phẩm PCR nhân bản đoạn gen trnL-trnF rất đặc hiệu, có thể sử dụng trực tiếp sản phẩm này để xác định trình tự nucleotide.

Hình 1. Kết quả điện di sản phẩm PCR trên gel Agarose
L: Ladder; 1 đến 18: băng sản phẩm PCR 18 mẫu trên gel
3.2. Kết quả phân tích trình tự
* Xác định trình tự ribosom vùng TrnL/TrnF
Kết quả giải trình tự đoạn gen trnL-trnF nhân bản từ 18 mẫu Quế cho thấy đoạn gen trnL-trnF phân lập từ 18 mẫu Quế có kích thước khoảng 390 bp và thành phần các nucleotid được thể hiện rõ và chi tiết tại Bảng 4.
Bảng 4: Thành phần bốn loại nucleotide của 18 mẫu Quế
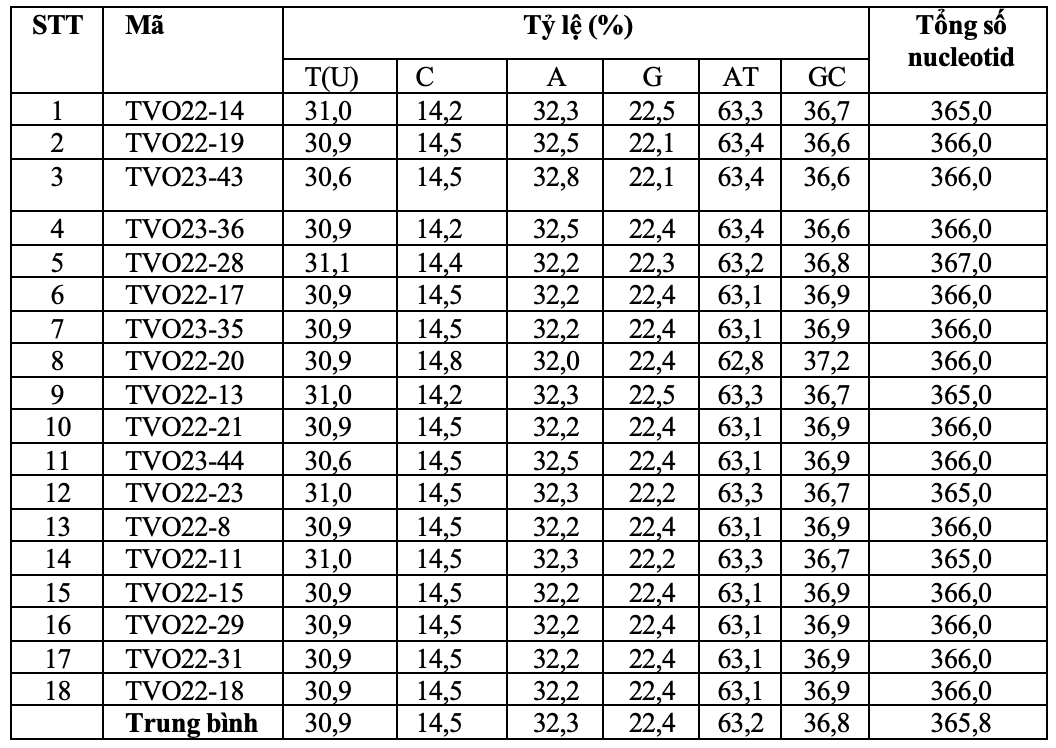
Sau khi khuếch đại đoạn gen tRNA-Leu bằng cặp mồi TrnL/TrnF, tất cả 18 mẫu Quế đều xuất hiện băng đơn với kích thước 390 bp, phù hợp với kích thước thường gặp ở nhiều loài thực vật. Phân tích trình tự gen TrnL/TrnF của 18 mẫu Quế cho kết quả trình tự trung bình gồm 63,2% Adenine (A) và Thymine (T), 36,8% Guanine (G) và Cytosine (C), cho thấy sự tương đồng cao giữa các mẫu. Điều này chứng minh đoạn gen TrnL/TrnF là một công cụ hiệu quả trong việc nhận diện và xác định mối quan hệ họ hàng giữa các mẫu quế (Bảng 4).
* So sánh trình tự DNA ribosom vùng TrnL/TrnF

Hình 2. Kết quả so sánh trình tự nucleotit với 18 mẫu Quế nghiên cứu
Sản phẩm PCR với cặp mồi TrnL/TrnF sau khi tinh sạch được phân tích trực tiếp trên máy giải trình tự ABI PRISM 3700 DNA Analyzer (Applied Biotech) của công ty TNHH DNA Sequecing – Việt Nam. Phần mềm MEGA v6.06 được sử dụng để phân tích và so sánh trình tự thu được với trình tự tham chiếu AB054230.1 của quế thật (Cinnamomum cassia) được công bố trên NCBI thông qua công cụ Blast (Basic Local Alignment Search Tool). Kết quả thu được 18 đoạn trình tự của 18 mẫu Quế nghiên cứu có sự tương đồng rất cao từ 99,46% đến 100%. Bước đầu với 18 mẫu lá Quế được thu thập từ các vùng khác nhau, nhóm nghiên cứu nhận thấy là cùng một loài (Cinnamomum cassia) (Hình 2).
Kết quả thu được khi so sánh trình tự nucleotit của 18 mẫu Quế nghiên cứu với trình tự tương ứng trên ngân hàng gen đã được công bố (AB054230.1) cho thấy sự tương đồng cao, phù hợp với kết quả khi so sánh trình tự nucleotid để tìm loài tương ứng thông qua Blast (http://blast.ncbi.nlm.nih.gov/Blast.cgi). Thực hiện so sánh từng cặp bằng công cụ BLAST, thu được hệ số tương đồng trình tự vùng TrnL/TrnF giữa 18 mẫu quế nghiên cứu. Kết quả ma trận nhận dạng trình tự được thể hiện ở bảng 5 (Bảng 5).
Bảng 5: Hệ số tương đồng và khoảng cách di truyền từ vùng TrnL/TrnF giữa 18 mẫu Quế
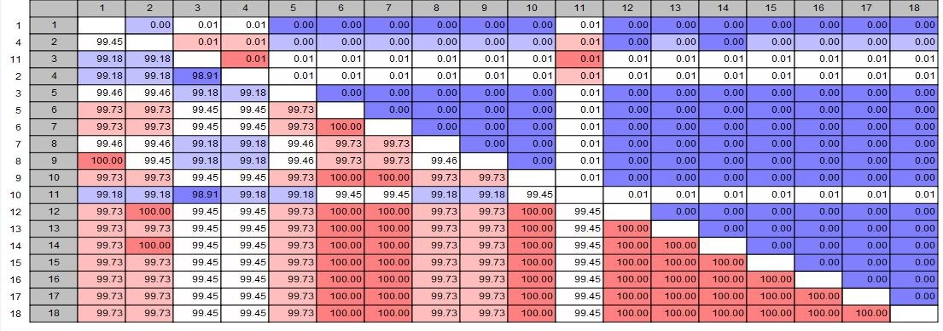
Kết quả trong bảng cho thấy có sự tương đồng cao giữa 18 trình tự của 18 mẫu quế hệ số tương đồng cao nhất là 100%, còn hệ số tương đồng thấp nhất là 98,91%. Và khoảng cách di truyền gần nhất là 0,00 và xa nhất là 0,01.
3.3. Xây dựng cây phát sinh loài
Sau khi xác định được trình tự nucleotid vùng TrnL/TrnF tiến hành dựng cây phân loại. Kết quả thể hiện ở (Hình 3).
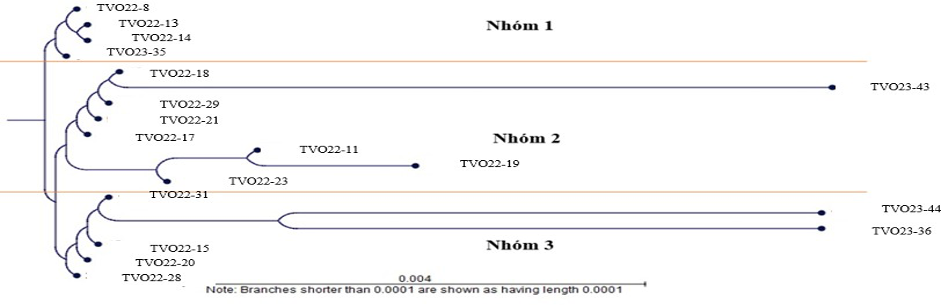
Hình 3. Cây phân loại của 18 mẫu Quế dựa trên so sánh trình tự TrnL/TrnF
Phân tích cây phát sinh loài bằng phần mềm Mega 6.06 theo phương pháp Maximum likelihood đã chia mẫu quế thành 3 nhóm chính:
Nhóm I: Bao gồm 4 mẫu nghiên cứu là mẫu TVO22-8 ở Bảo Thắng Lào Cai, TVO22-13 và TVO22-14 ở Cao Sơn, Đà Bắc (Hòa Bình),mẫu TVO23-35 Chợ Đồn (Bắc Kạn). Với độ tương đồng trình tự từ 99,73% đến 100%. Bốn mẫu này có hệ số tương đồng về trình tự nucleotid với mẫu tham chiếu Cinnamomum cassia khoảng 99,73% đến 100%.
Nhóm II: Chiếm tỷ lệ cao nhất bao gồm 8 mẫu, chia thành 2 nhóm nhỏ.
Nhóm II.1: Gồm 5 mẫu nghiên cứu TVO22-17, TVO22-18 Hoành Mô, Quảng Ninh; TVO22-21 Thường Xuân (Thanh Hóa); TVO22-29 Đà Bắc (Hòa Bình); TVO23- 43 Bắc Hà (Lào Cai) , với độ tương đồng khoảng 99,18% đến 100% so sánh với mẫu tham chiếu Cinnamomum cassia có độ tương đồng 99,73% đến 100%. Trong đó độ tường đồng thấp nhất ở mẫu TVO23-43 các mẫu còn lại độ tương đồng 100%.
Nhóm II.2: gồm 3 mẫu TVO22-23 Trà Mai (Quảng Nam); TVO22-19 Hoành Mô (Quảng Ninh); TVO22-11 Đại Sơn (Yên Bái); nhóm có hệ số tương đồng 98,91 đến 100%, so với mẫu tham chiếu Cinnamomum cassia có độ tương đồng 100%.
Nhóm III. Gồm có 6 mẫu có TVO22-31Trung Thành (Hòa Bình) TVO23-44 Nậm Đét (Lào Cai); TVO23-36 Nà Ngà (Bắc Kạn); TVO22-15 Cao Sơn (Hòa Bình); TVO22-20 Luân Thành (Thanh Hóa); TVO22-28 Làng Muông (Thái Nguyên); các mẫu có độ tương đồng từ 99,18% đến 100%, so với mẫu tham chiếu Cinnamomum cassia có độ tương đồng 99,46% đến 100%.
4. Kết luận
Kết quả PCR khuếch đại vùng TrnL/TrnF ở 18 mẫu Quế ở vị trí băng AND xấp xỉ 400bp, các băng DNA, không lẫn tạp chất, tạo tiền đề vững chắc cho quá trình giải trình tự gen. Dự trên cây phát sinh loài, 18 mẫu giống Quế chia thành 3 nhóm chính và có hệ số tương đồng cao (xấp xỉ 99%). Phân tích trình tự nucleotid đã cung cấp thông tin di truyền quan trọng, hỗ trợ xác định và so sánh đặc điểm di truyền của các mẫu quế từ các vùng sinh thái khác nhau. Nghiên cứu không chỉ góp phần vào việc bảo tồn nguồn gen quế quý hiếm mà còn mở ra hướng ứng dụng tiềm năng trong việc phát triển và tối ưu hóa các giống quế chất lượng cao phục vụ ngành dược liệu và công nghiệp chế biến.
Tài liệu tham khảo
1. Võ Văn Chí (2003), Từ điển Thực vật thông dụng, Tập 1, NXB Khoa học và Kỹ thuật, Hà Nội, tr. 669-679.
2. Phạm Xuân Hoàn (2001), Nghiên cứu sinh trưởng và sản lượng làm cơ sở đề xuất một số biện pháp kỹ thuật trong kinh doanh rừng Quế (Cinnamomum cassia ) tại tỉnh Yên Bái), Tóm tắt luận án tiến sỹ nông nghiệp, Trường Đại học lâm nghiệp, Hà Tây.
3. Nguyễn Thị Hà (2021), Hiện trạng nghiên cứu, sử dụng và tiềm năng phát triển của phân bón sinh học ở Châu Á và Việt Nam, NXB Nông Nghiệp Việt Nam.
4. Balijepalli, Madhu Katyayani, Ayuba Sunday Buru, Raghavendra Sakirolla, Mallikarjuna Rao Píchika. (2017), "Cinnamomum genus: A review on its biological activities", International Journal of Pharmacy Pharmaceutical Sciences 9(2), pp. 1-11.
5. Takhtajan A. (2009), Flowering plants, Springer.
6. Phạm Hoàng Hộ (1999), Cây cỏ Việt Nam, Quyển 1, NXB trẻ, Hà Nội, tr.343- 351
7. AM Sandigawad, CG Patil (2011), "Genetic diversity in some south-Indian Cinnamomum Scha. species revealed by RAPD markers", Indian Journal of Genetics Plant Breeding 71(01), pp. 87-90.
8. PD Abeysinghe, KGG Wijesinghe, H Yoshda, T Tachida. (2009), "Molecular characterization of Cinnamon (Cinnamomum verum Presl) accessions and evaluation of genetic relatedness of Cinnamon species in Sri Lanka based on trnL intron region, intergenic spacers between trnT-trnL, trnL-trnF, trnH-psbA and nuclear ITS", Research Journal of Agriculture and Biological Sciences, 5(6), pp. 1079-1088.
9. KY Ho, TY Hung (2011), "Cladistic relationships within the genus Cinnamomum (Lauraceae) in Taiwan based on analysis of leaf morphology and inter-simple sequence repeat (ISSR) and internal transcribed spacer (ITS) molecular markers", African Journal of Biotechnology (AJB).
10. Khuất Hữu Trung, Kiều Thị Dung, Lưu Cảnh Trung, Nguyễn Huy Hoàng, Hồ Trung Lương (2017), “Đánh giá đa dạng di truyền các loài quế Thanh Hóa bằng chỉ thị RAPD”, Tạp Chí Khoa học Và Công nghệ Việt Nam, 59(2), tr.1-5.
11. Pushpa, Divya Prasad, Smita Hemrom, Masroor Jahan, A. N. Verma, Amool R. Singh (2009), "Efficacy of psycho-education with the patients of schizophrenia", Eastern Journal of Psychiatry 12(1&), p. 37.
12. C Zhang, L Fan, S Fan, J Wang, T Luo, Y Tang, Z Chen (2019), "Cinnamomum cassia Presl: A Review of Its Traditional Uses, Phytochemistry, Pharmacology and Toxicology, " Molecules, 24(19), p. 3473.
13. R Azad, KLW Kumara, G Senanayake, et al. (2018), "Flower morphological diversity of cinnamon (Cinnamomum verum Presl) in Matara district, Sri Lanka", Open Agriculture 3(1), pp. 236-244.
14. PN Kaul, AK Bhattacharya, et al. (2003), "Volatile constituents of essential oils isolated from different parts of cinnamon (Cinnamomum zeylanicum Blume)", Journal of the Science of Food Agriculture 83(1), pp. 53-55.
MOLECULAR DIVERSITY AND IDENTIFICATION OFCICINNAMON ( Cinamomum) SPECIES BY USING TrnL/TrnF SEQUENCING
Abstract:
The genus Cinnamomum, comprising approximately 250 species globally, is a plant group with significant applications in medicine. In Vietnam, previous studies have identified three primary cinnamon species: Cinnamomum cassia, Cinnamoum loureirii, and Cinnamomum zeylanicum. However, these classifications remain contentious, primarily due to their reliance on morphological and chemical characteristics. To elucidate the genetic diversity of Vietnamese cinnamon species, we conducted a sequence analysis of the trnL/trnF intergenic spacer region on 18 samples collected from four distinct ecological zones across Vietnam (Northeast, Northwest, North Central Coast, and South Central Coast). The selected samples were mature trees (10-15 years old), representing the geographic diversity of Vietnamese cinnamon. The nucleotide sequence analysis of the trnL/trnF region provided a more in-depth understanding of the genetic relationships among the samples. A high level of genetic similarity was observed among the samples, with an average A+T content of 63.2% and G+C content of 36.8%. Sequence similarity coefficients among the samples ranged from 98.91% to 100%, and comparisons with database sequences confirmed that all samples belonged to the species Cinnamomum cassia."
Keywords: Cinnamomum, trnL/trnF, DNA, genetic diversity
Nguyễn Thu Hiền - Trường Đại học Dược Hà Nội
Nguyễn Thị Nguyệt - Trường Đại Học Tân Trào
Bùi Thị Kim Oanh - Trường Đại học Dược Hà Nội
Bùi Thị Lan Phương - Trường Đại học Dược Hà Nội
Nguyễn Thị Kiều Anh - Trường Đại học Dược Hà Nội
Trần Văn Ơn - Trường Đại học Dược Hà Nội
Nguyễn Trường Khoa - Viện Di truyền Nông Nghiệp
Trần Duy Cường - Viện Di truyền Nông Nghiệp