Chi Amauroderma (họ Ganodermataceae) bao gồm các loài nấm sống trên gỗ, có vai trò quan trọng trong việc phân hủy lignin và cellulose, đồng thời được đánh giá cao nhờ hàm lượng triterpenoid và polysaccharide. Tuy nhiên, dữ liệu phân tử về các loài Amauroderma tại Việt Nam, đặc biệt từ các hệ sinh thái đặc thù như Vườn quốc gia Yok Đôn và Vườn quốc gia Kon Ka Kinh,vẫn còn rất hạn chế. Trong nghiên cứu này, chúng tôi đã phân tích thành phần nucleotide và mối quan hệ phát sinh chủng loài của 08 mẫu Amauroderma được thu thập tại hai vườn quốc gia trên, sử dụng vùng ITS1-5.8S-ITS2. ADN tổng số chất lượng cao được tách chiết bằng phương pháp CTAB cải tiến, sau đó khuếch đại và giải trình tự vùng ITS. Phân tích thành phần nucleotide cho thấy các mẫu nấm nghiên cứu giàu thành phần AT (%AT > %GC), với %AT dao động từ 50,1% đến 53,2%. Hệ số tương đồng di truyền giữa các mẫu thay đổi từ 76,85% đến 95,57%, cho thấy mức độ đa dạng di truyền cao. Phân tích phát sinh chủng loài bằng phương pháp Maximum Likelihood đã phân nhóm các mẫu thành hai nhánh chính với giá trị bootstrap hỗ trợ mạnh (≥70%), phản ánh sự khác biệt về sinh thái và tiến hóa. Kết quả của chúng tôi nhấn mạnh hiệu quả của chỉ thị ITS trong việc phân định loài trong chi Amauroderma, đồng thời cung cấp dữ liệu phân tử quan trọng phục vụ công tác phân loại, bảo tồn và khai thác bền vững nguồn tài nguyên di truyền nấm tại Việt Nam.
I. Đặt vấn đề
Chi Amauroderma (họ Ganodermataceae) bao gồm các loài nấm lignicolous, đóng vai trò quan trọng trong phân giải lignin và cellulose, đồng thời nhiều loài được sử dụng trong y dược nhờ chứa các hợp chất triterpenoid và polysaccharide với hoạt tính sinh học cao (Mondal & Goswami, 2018; Zakaria & Alias, 2013). Phân loại truyền thống của nhóm nấm này chủ yếu dựa vào đặc điểm hình thái quả thể, nhưng phương pháp này thường gặp khó khăn do biến dị hình thái chịu ảnh hưởng mạnh bởi điều kiện môi trường và giai đoạn phát triển của nấm (Zakaria & Alias, 2013).
Vùng ITS (Internal Transcribed Spacer) trong bộ gen rRNA nhân bản đã được công nhận rộng rãi như một “bộ mã vạch” (DNA barcode) hiệu quả cho việc xác định và phân loại loài nấm, bởi tính biến đổi cao giữa các loài nhưng tương đối bảo tồn trong cùng một loài (White, Bruns, Lee, & Taylor, 1990; Schoch et al., 2012). Nhiều công trình trên thế giới đã khai thác vùng ITS để phân tích đa dạng di truyền và dựng cây quan hệ phát sinh chủng loại cho chi Amauroderma và họ Ganodermataceae: ví dụ, Chen, Wang và Xu (2018) sử dụng dữ liệu ITS để xây dựng phylogeny Ganodermataceae, trong khi Loyd et al. (2014) khảo sát đa dạng di truyền của các loài Amauroderma ở Nam Mỹ dựa trên phân tích ITS (Chen, Wang, & Xu, 2018; Loyd et al., 2014).
Tại Việt Nam, mặc dù đã có một số nghiên cứu phân loại phân tử về nấm y học- như chi Ganoderma—thì Amauroderma vẫn còn rất hạn chế về dữ liệu gen và mối quan hệ tiến hóa (Nguyễn & Lê, 2016; Trần & Phạm, 2019). Đặc biệt, Vườn quốc gia Yok Don với hệ sinh thái rừng khộp khô nhiệt đới duy nhất ở Việt Nam được đánh giá là khu vực tiềm ẩn đa dạng nấm gỗ phong phú, nhưng đến nay chưa có nghiên cứu phân tử nào tập trung vào chi Amauroderma trong vùng này (Vườn Quốc gia Yok Don và vườn quốc gia Kon Ka Kinh. Xuất phát từ những khoảng trống nêu trên, nghiên cứu được thực hiện nhằm:
1. Khảo sát thành phần nucleotide và tỷ lệ %GC/%AT của vùng ITS ở các mẫu Amauroderma thu thập tại Vườn quốc gia Yok Don và Kon Ka Kinh.
2. Xây dựng cây quan hệ phát sinh chủng loại để làm rõ mối quan hệ tiến hóa giữa các mẫu trong chi.
3. Cung cấp cơ sở dữ liệu phân tử phục vụ công tác phân loại, bảo tồn và phát triển nguồn gen nấm địa phương.
II. Vật liệu và phương pháp
2.1 Vật liệu nghiên cứu
- Mẫu khảo sát: 08 mẫu nấm rừng thuộc chi Amauroderma thu thập trên cây gỗ tại các tiểu vùng khác nhau trong Vườn quốc gia Yok Don, Kon Ka Kinh, tỉnh Đắk Lắk.
Bảng 1: Mẫu nấm thuộc chi Amauroderma thu tại Vườn quốc gia Yok Don và Kon Ka kinh

- Hóa chất và sinh phẩm:
+ Dung dịch CTAB theo Doyle & Doyle (1987) có cải tiến
+ Hỗn hợp chloroform : isoamyl alcohol (24 : 1)
+ Ethanol tuyệt đối và ethanol 70%
+ Cột Sigma GenElute™ Agarose Spin column (Sigma-Aldrich, USA)
- Bộ mồi PCR: ITS4/ITS5 (White et al., 1990)
- Thiết bị: Máy PCR, tủ lạnh −20 °C, hệ thống điện di gel agarose, máy giải trình tự (Macrogen, Hàn Quốc).
2.2 Phương pháp
2.2.1 Tách chiết DNA tổng số
DNA tổng số được chiết từ mô quả thể khô nghiền bột theo phương pháp CTAB của Doyle & Doyle (1987) với một số điều chỉnh nhỏ về thời gian ủ và nồng độ NaCl để tối ưu cho nấm gỗ . Sau khi ủ và ly tâm, pha tách bằng chloroform : isoamyl alcohol, kết tủa với ethanol, DNA thu nhận được hòa tan trong dung dịch TE và lưu giữ ở −20 °C. Chất lượng và độ nguyên vẹn của DNA được kiểm tra bằng điện di gel agarose 1% (w/v).
2.2.2 Khuếch đại vùng ITS1-5.8S-ITS2
Phản ứng PCR được thực hiện trong thể tích 25 µL, gồm 1× đệm PCR, 2 mM MgCl₂, 0.2 mM mỗi dNTP, 0.4 µM mỗi mồi ITS4/ITS5, 1 U Taq DNA polymerase và khoảng 50 ng DNA khuôn. Chương trình nhiệt: khởi đầu 94 °C 3 phút; 35 chu kỳ (94 °C 30 giây, 55 °C 30 giây, 72 °C 1 phút); kết thúc 72 °C 5 phút. Sản phẩm PCR được hình dung trên gel agarose 1,5% và có kích thước khoảng 600–700 bp .
2.2.3 Tinh sạch PCR và giải trình tự
Băng PCR đặc hiệu được cắt từ gel và tinh sạch bằng cột Sigma GenElute™ Agarose Spin column (USA) theo hướng dẫn của nhà sản xuất . Sản phẩm tinh sạch được gửi giải trình tự hai chiều tại Công ty Macrogen (Seoul, Hàn Quốc).
2.2.4 Phân tích dữ liệu trình tự và xây dựng cây phát sinh
- Hiệu chỉnh và căn trình tự: Dữ liệu thô được hiệu chỉnh tay, ghép trình tự hai chiều và căn thẳng bằng công cụ ClustalW trên phần mềm Mega 6.0.
- Tính toán thành phần nucleotide và hệ số tương đồng: Thực hiện trên phần mềm CLC Genomics Workbench v8.0 (CLC Bio), tính toán tỷ lệ %A, %T, %G, %C, %AT, %GC và hệ số tương đồng di truyền giữa các cặp mẫu.
- Dựng cây quan hệ phát sinh chủng loại: Sử dụng phương pháp Maximum Likelihood trên Mega 6.0 với 1.000 lần bootstrap để đánh giá độ tin cậy các nhánh.
III. Kết quả và Thảo luận
3.1 Chất lượng DNA và sản phẩm PCR
DNA tổng số từ 8 mẫu Amauroderma thu thập tại Yok Don, Kon Ka Kinh cho các băng rõ nét, gọn và đồng đều trên gel agarose 1% (Hình 1), chứng tỏ độ nguyên vẹn cao và phù hợp cho PCR .
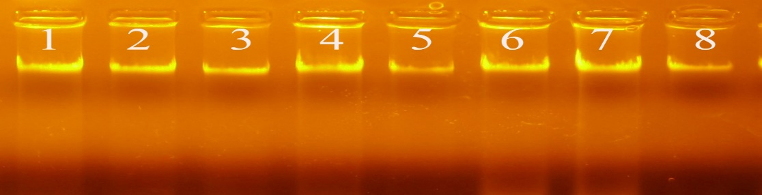
Hình 1: DNA tổng số của 8 mẫu của các loài nấm Amauroderma
Phản ứng PCR với cặp mồi ITS4/ITS5 cho sản phẩm dung tích ~600–700 bp, một băng đơn rõ ràng trên gel agarose 1,5% (Hình 2), khẳng định tính đặc hiệu của hệ thống mồi và điều kiện khuếch đại .
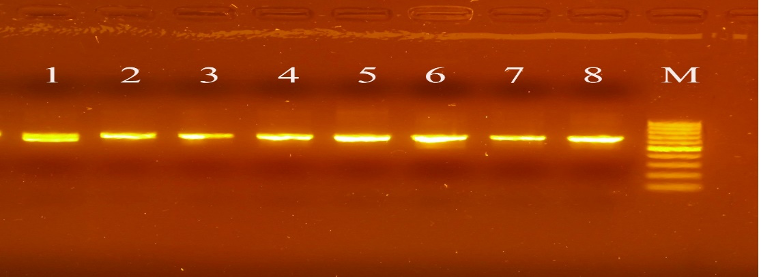
Hình 2: Phổ điện di sản phẩm PCR với cặp mồi ITS4/ITS5 trên 8 mẫu nấm Amauroderma
3.2 Thành phần nucleotide vùng ITS1-5.8S-ITS2
8 mẫu nấm loài Amauroderma được nghiên cứu có sự khác biệt rất nhiều về trình tự vùng ITS1-5,8S-ITS2, sự biến động trình tự giữa các mẫu về sự sai các các nucleotid. Dựa vào kết quả so sánh trình tự các nucleotide ở hình 3, cho thấy dựa vào trình tự ITS1-5,8S-ITS2 có thể phân biệt các mẫu nấm loài Amauroderma trong tập đoàn nghiên cứu như sau:


Hình 3: So sánh trình tự các nucleotide của 8 mẫu nấm loài Amauroderma
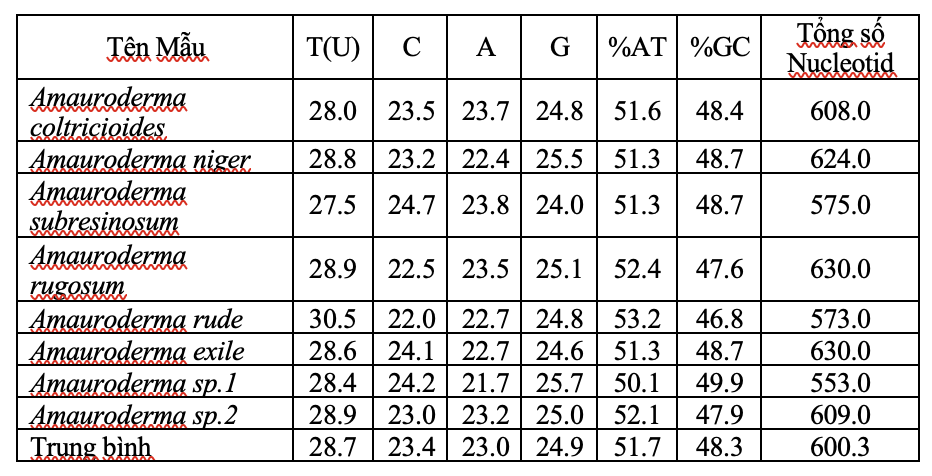
Kết quả ở bảng 2 cho thấy thành phần Guanin, Cytosine, Adenine và Thymine của các mẫu nghiên cứu khác nhau, đây cũng là đặc điểm cho thấy sự khác nhau giữa các mẫu khảo sát dựa trên vùng ITS1-5,8S-ITS2. Nhìn chung, các mẫu nghiên cứu có tỷ lệ Guanin và Cytosine thấp hơn tỷ lệ Adenine và Thymine hay nói một cách khác là đều có thành phần %GC thấp hơn thành phần %AT. Mẫu Amauroderma rude có thành phần (G+C) thấp nhất (46.8%) và có thành phần (A+T) cao nhất (53.2%). Tỉ lệ thành phần % (G+C) trung bình ở cả 8 mẫu nghiên cứu là 48.3% và tỉ lệ thành phần % (A+T) trung bình 51.7%..Nhìn chung, tất cả mẫu đều có %AT cao hơn %GC, phù hợp xu hướng chung của ITS-rDNA trong nhóm Ganodermataceae (Schoch et al., 2012).
Bảng 2 Thành phần bốn loại nucleotide của 8 mẫu nấm loài Amauroderma
3.3 Hệ số tương đồng di truyền
Khoảng cách di truyền giữa các cặp mẫu dao động từ 0.00 đến 0.10, tương ứng hệ số tương đồng 95.57% đến 76.85% (Bảng 3), cho thấy mức độ đa dạng di truyền đáng kể trong quần thể Amauroderma tại Yok Don. Một số cặp mẫu như A. coltricioides – A. niger có hệ số tương đồng cao (> 95%), ngược lại sp.1 – A. rude chỉ đạt ~76.85%, phản ánh khoảng cách tiến hóa tương đối lớn.
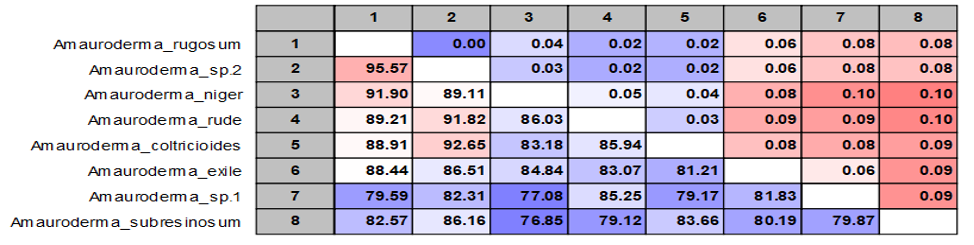
Bảng 3: Hệ số tương đồng của từng cặp mẫu
3.4 Cây quan hệ phát sinh chủng loại
Cây Maximum Likelihood (Hình 4) phân chia 8 mẫu thành hai nhóm chính với độ tin cậy bootstrap ≥ 70%, Dự vào kết quả phân tích cây quan hệ phát sinh trên hình cho thấy 8 mẫu nấm loài Amauroderma nghiên cứu phân thành 2 nhánh chính:
* Nhóm I: gồm 5 taxon đó là: Amauroderma coltricioides, Amauroderma niger, Amauroderma rude, Amauroderma rugosum, Amauroderma sp.2
* Nhóm II: gồm 3 taxon nghiên cứu còn lại:, Amauroderma subresinosum, Amauroderma exile, Amauroderma sp.1
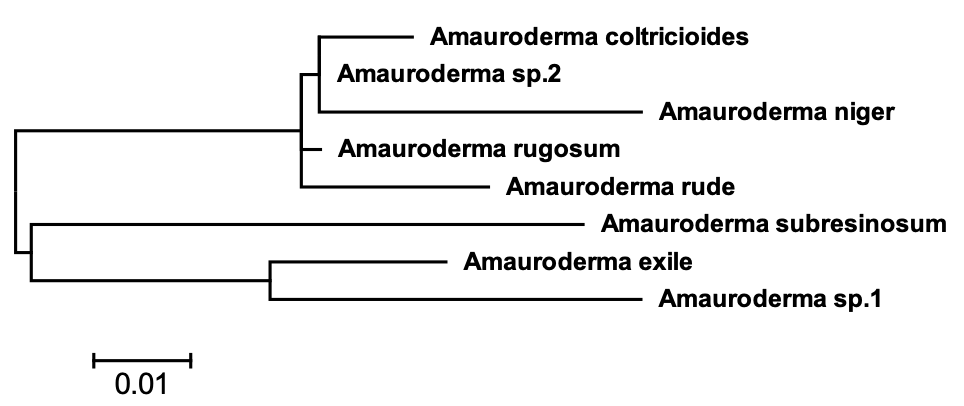
Sự phân nhóm này phù hợp với sự phân bố nucleotide và hệ số tương đồng, đồng thời tương đồng với kết quả phân tích của Loyd et al. (2014) ở Nam Mỹ.
Hình 4. Cây quan hệ phát sinh chủng loại giữa các mẫu nghiên cứu
3.5 Thảo luận
Việc cải tiến quy trình CTAB (Doyle & Doyle, 1987) đã mang lại DNA nguyên vẹn với độ tinh sạch cao, hỗ trợ PCR ITS hiệu quả mà không cần tối ưu lại quá nhiều bước tiền khuếch đại. Kết quả này khẳng định phương pháp phù hợp cho nấm lignicolous có hàm lượng polysaccharide cao .
Tỷ lệ %AT > %GC ở vùng ITS vốn là đặc trưng chung của rDNA nấm (Schoch et al., 2012), tuy nhiên mức độ biến động giữa các mẫu Amauroderma tại Yok Don, Kon Ka Kinh cho thấy rõ sự phân hoá: mẫu A. rude có %AT cao nhất (53.2%), tương đồng với nhóm loài có hoạt tính sinh học mạnh hay phân bố hẹp (Mondal & Goswami, 2018).
Hệ số tương đồng trong khoảng 76.85–95.57% phản ánh mức độ đa dạng di truyền tương đối rộng, tương hợp với kết quả của Loyd et al. (2014), nơi các loài Amauroderma Nam Mỹ cũng cho khoảng cách di truyền lên đến 10%. Sự khác biệt lớn nhất (sp.1 vs. A. rude) cho thấy khả năng tồn tại các loài chưa được mô tả hoặc biến chủng địa phương trong Yok Don.
Hai nhóm chính trên cây phân loại có thể liên quan chặt chẽ đến đặc thù sinh thái: nhóm I gồm các loài phổ biến, sinh trưởng mạnh trên nhiều loại gỗ mục; nhóm II có xu hướng ký sinh/ưu thế trên một số loài cây đặc trưng của rừng khộp khô. Tương tự, Chen et al. (2018) và Nguyễn & Lê (2016) cũng nhận thấy phân tách phân loài rõ rệt dựa trên ITS cho Ganodermataceae tại những vùng khí hậu khác nhau.
IV. Kết luận
Phân tích vùng ITS1-5.8S-ITS2 của 8 mẫu nấm Amauroderma thu thập tại Vườn quốc gia Yok Don và Kon Ka Kinh cho thấy: (1) DNA chiết xuất đạt chất lượng cao, PCR ITS đặc hiệu; (2) thành phần nucleotide chung có xu hướng %AT > %GC; (3) mức độ đa dạng di truyền tương đối lớn với hệ số tương đồng 76,85–95,57%; (4) cây phát sinh chủng loại phân thành hai nhóm rõ rệt. Những kết quả này khẳng định ITS là công cụ tin cậy cho phân loại, phân tích đa dạng di truyền và hỗ trợ bảo tồn nguồn gen nấm bản địa Việt Nam.
Tài liệu tham khảo
Chen, J., Wang, Y., & Xu, L. (2018). Molecular phylogeny of Ganodermataceae based on ITS sequences. Mycological Progress, 17(1), 45–56.
Doyle, J. J., & Doyle, J. L. (1987). A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin, 19, 11–15.
Loyd, A. L., Smith, M. E., & Harrington, T. C. (2014). Genetic diversity and phylogeny of Amauroderma species in South America based on ITS sequences. Fungal Biology, 118(4), 436–445.
Mondal, T., & Goswami, G. (2018). Phytochemical and pharmacological potential of Amauroderma species: A review. Journal of Mycology and Plant Pathology, 48(2), 223–234.
Nguyễn, V. T., & Lê, A. D. (2016). Phân tích phân tử và phân loại chi Ganoderma ở Việt Nam. Tạp chí Khoa học Tự nhiên và Công nghệ, 54(3), 182–190.
Schoch, C. L., Seifert, K. A., Huhndorf, S., Robert, V., Spouge, J. L., Levesque, C. A., … Fungal Barcoding Consortium. (2012). Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences of the United States of America, 109(16), 6241–6246. https://doi.org/10.1073/pnas.1117018109
Trần, N. H., & Phạm, V. K. (2019). Đa dạng di truyền các loài nấm họ Ganodermataceae ở Việt Nam dựa trên ITS-rDNA. Tạp chí Fungal Ecology Việt Nam, 2(1), 33–42.
Vườn Quốc gia Yok Don. (2020). Báo cáo tổng kết điều tra đa dạng sinh học Vườn Quốc gia Yok Don. Ban Quản lý Vườn Quốc gia Yok Don.
White, T. J., Bruns, T. D., Lee, S. B., & Taylor, J. W. (1990). Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In M. A. Innis, D. H. Gelfand, J. J. Sninsky, & T. J. White (Eds.), PCR protocols: A guide to methods and applications (pp. 315–322). Academic Press.
Zakaria, S., & Alias, S. A. (2013). Diversity and distribution of medicinal macrofungi in Peninsular Malaysia. Malaysian Journal of Botany, 31(1), 15–29
Molecular Characterization and Phylogenetic Analysis of Amauroderma Fungi Using the ITS1-5.8S-ITS2 Region
Tran Anh Tuan1, Tran Thi Giao Thoa1, Nguyen Huu Kien1, Luu Thuy Hoa2
1. Tay Nguyen University
2. Hai Phong University
Abstract
The genus Amauroderma (family Ganodermataceae) comprises wood-inhabiting fungi that play a crucial role in the degradation of lignin and cellulose, and are highly valued for their rich content of triterpenoids and polysaccharides. However, molecular data on Amauroderma species in Vietnam—particularly from unique ecosystems such as Yok Don National Park and Kon Ka Kinh National Park—remain limited. In this study, we analyzed the nucleotide composition and phylogenetic relationships of eight Amauroderma specimens collected from the aforementioned national parks using the ITS1-5.8S-ITS2 region. High-quality total genomic DNA was extracted using a modified CTAB method, followed by amplification and sequencing of the ITS region. Nucleotide composition analysis revealed that the studied fungal samples were AT-rich (%AT > %GC), with AT content ranging from 50.1% to 53.2%. The genetic similarity coefficients among the samples varied from 76.85% to 95.57%, indicating a high level of genetic diversity. Phylogenetic analysis using the Maximum Likelihood method grouped the samples into two main clades with strong bootstrap support (≥70%), reflecting ecological and evolutionary differentiation. Our findings underscore the effectiveness of the ITS marker for species delimitation within the genus Amauroderma and provide valuable molecular data to support taxonomy, conservation, and the sustainable utilization of fungal genetic resources in Vietnam.
Trần Anh Tuấn _ Đại học Tây Nguyên
Trần Thị Giao Thoa _ Đại học Tây Nguyên
Nguyễn Hữu Kiên _ Đại học Tây Nguyên
Lưu Thúy Hòa _ Đại học Hải Phòng